微生物多样性测序是基于第二代高通量技术对16S rRNA/18S rRNA/ITS等基因序列进行测序,能同时对样品中的优势物种、稀有物种以及一些未知的物种进行检测,获得样品中的微生物群落组成以及它们之间的相对丰度。探讨微生物多样性对于研究微生物与环境的关系、环境治理和微生物资源的利用有着重要的理论和现实意义。
16S rDNA测序: 16SrDNA为编码原核生物核糖体小亚基rRNA的DNA序列,具有10个保守区域和9个高变区域,其中保守区在细菌中差异不大,高变区具有属和种的特异性,对16S rDNA某个高变区进行测序,用于研究环境微生物中细菌或古菌的群落多样性。
16S rDNA测序: 16SrDNA为编码原核生物核糖体小亚基rRNA的DNA序列,具有10个保守区域和9个高变区域,其中保守区在细菌中差异不大,高变区具有属和种的特异性,对16S rDNA某个高变区进行测序,用于研究环境微生物中细菌或古菌的群落多样性。
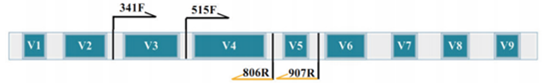
18S rDNA测序: 18S rDNA为编码真核生物核糖体小亚基rRNA的DNA序列,对18S rDNA某个高变区进行测序,用于研究环境微生物中真核微生物的群落多样性。
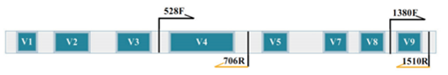
ITS测序: ITS分为两个区域ITS1h和ITS2,ITS1位于真核生物rDNA序列18S和5.8S之间,ITS2位于真核生物 rDNA序列5.8S和28S之间。对ITS1或ITS2进行测序,用于研究环境微生物中真菌群落结构多样性。
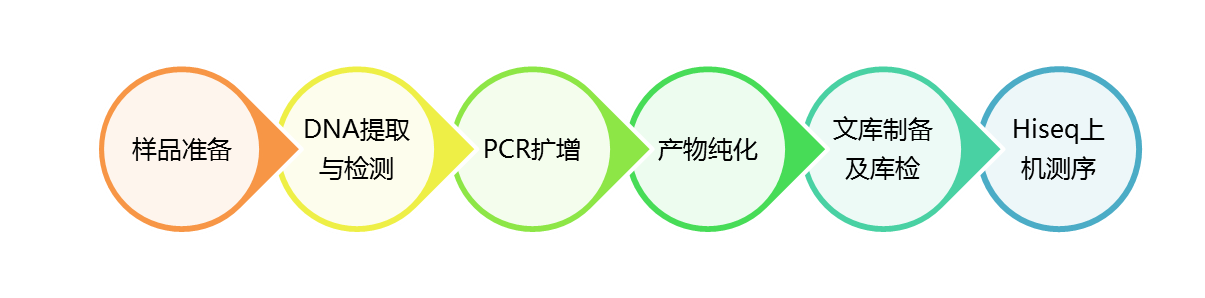
技术流程
技术优势
● 灵活的研究方案:针对客户的不同研究需求提供灵活多变的研究方案
● 专业严谨的服务:专业成熟的样本制备、建库以及数据分析流程
● 专业的数据分析能力:针对大量样本及分组复杂样本,具有专业的分析能力
分析流程
● 灵活的研究方案:针对客户的不同研究需求提供灵活多变的研究方案
● 专业严谨的服务:专业成熟的样本制备、建库以及数据分析流程
● 专业的数据分析能力:针对大量样本及分组复杂样本,具有专业的分析能力
分析流程
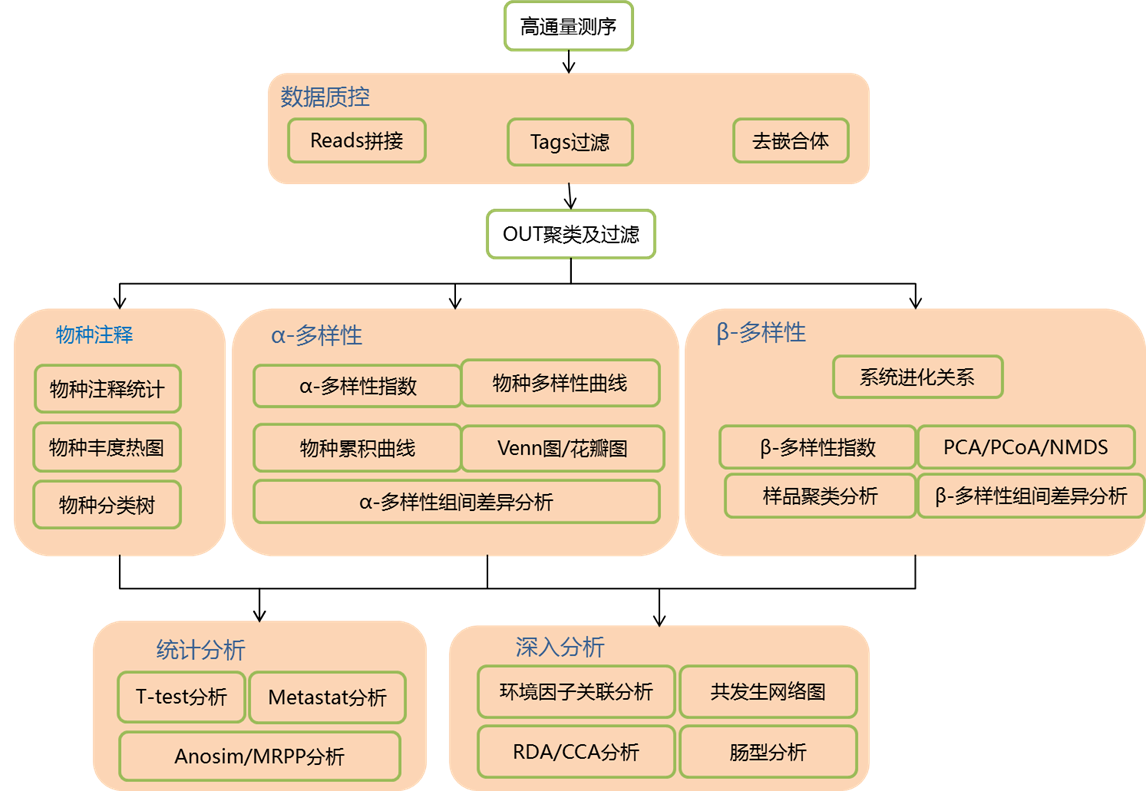
结果展示(部分)
● 测序数据处理
采用 Illumina HiSeq 测序平台得到的下机数据,我们称之为Raw PE,然后进行拼接和质控,得到Clean Tags,再进行嵌合体过滤,得到可用于后续分析的有效数据,即Effective Tags。
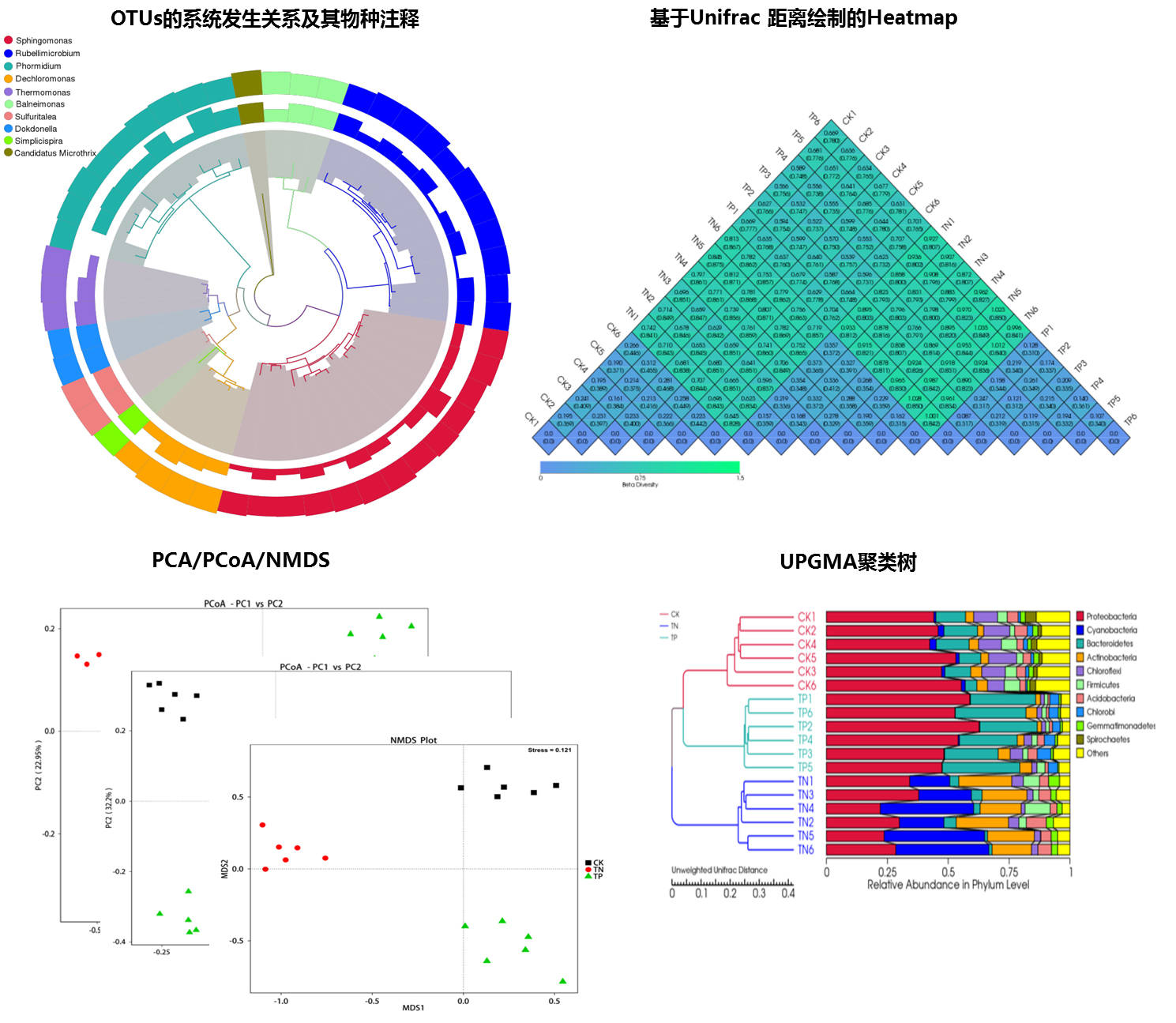
● OTU分析和物种注释
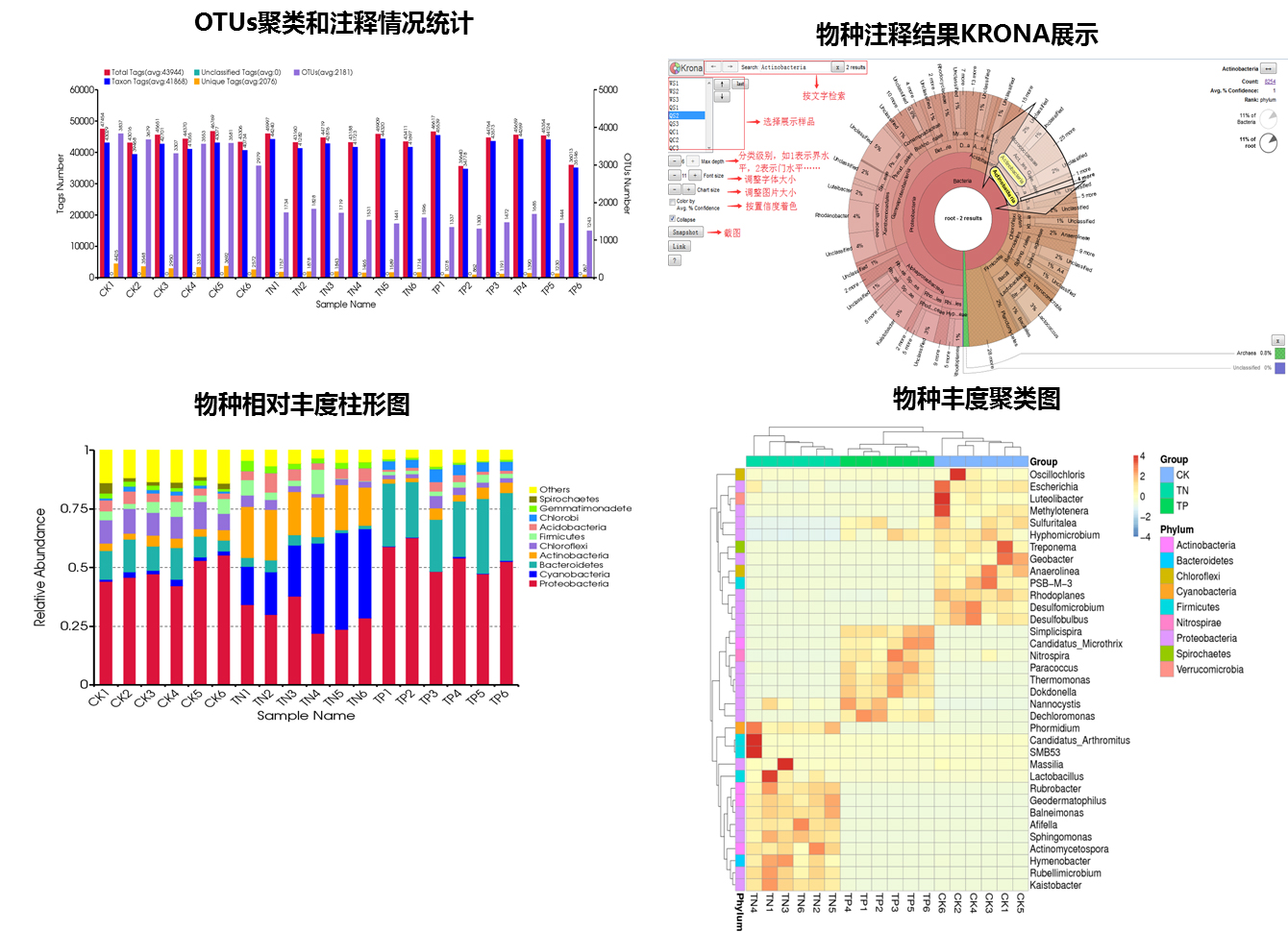
●样品复杂度分析(Alpha Diversity)
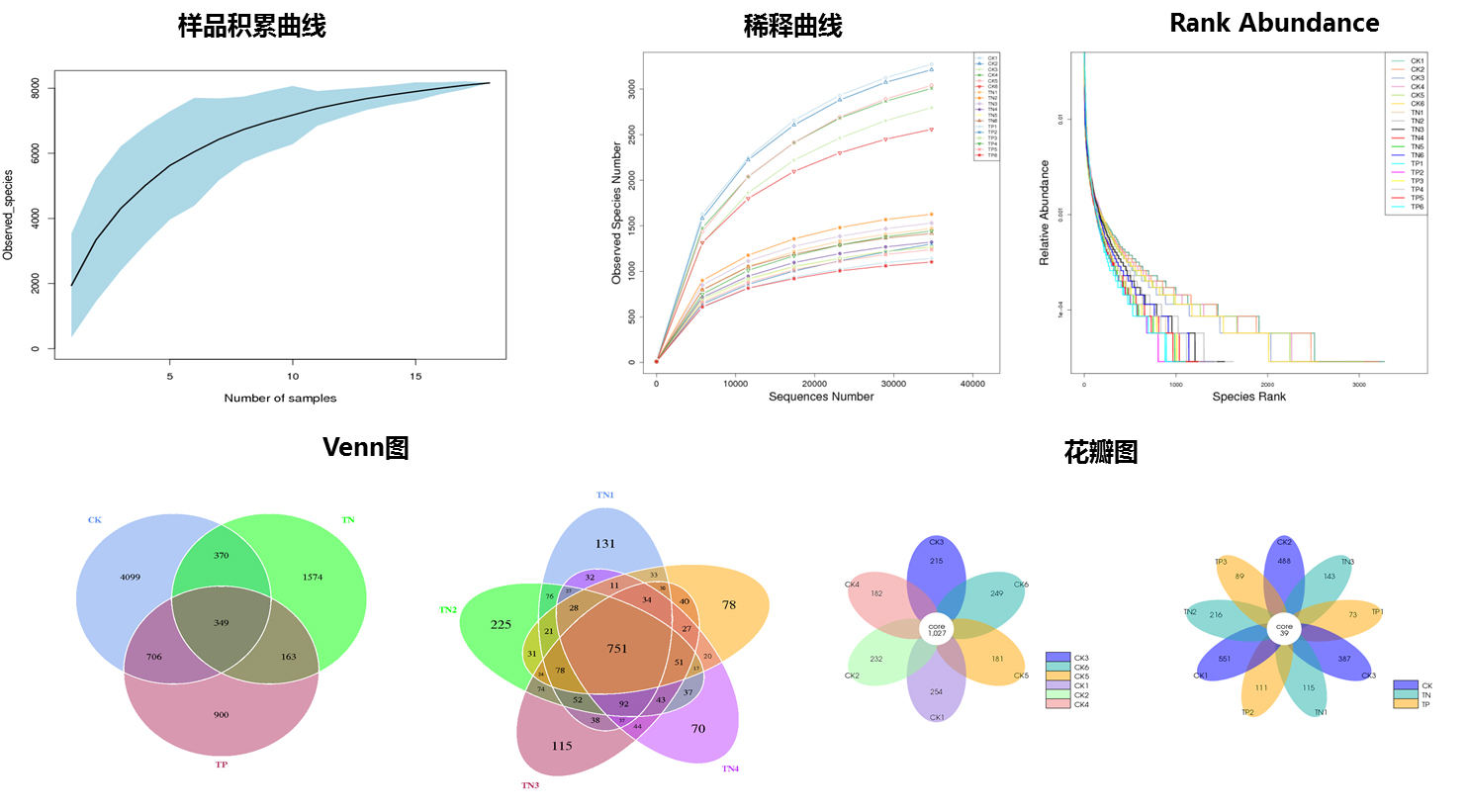
Alpha Diversity 用于分析样品内微生物群落多样性,通过物种累积曲线、物种多样性曲线和一系列统计学分析指数来评估各样品中微生物群落的物种丰富度和多样性的差异。(用于评估样本内物种丰富度和均一性)
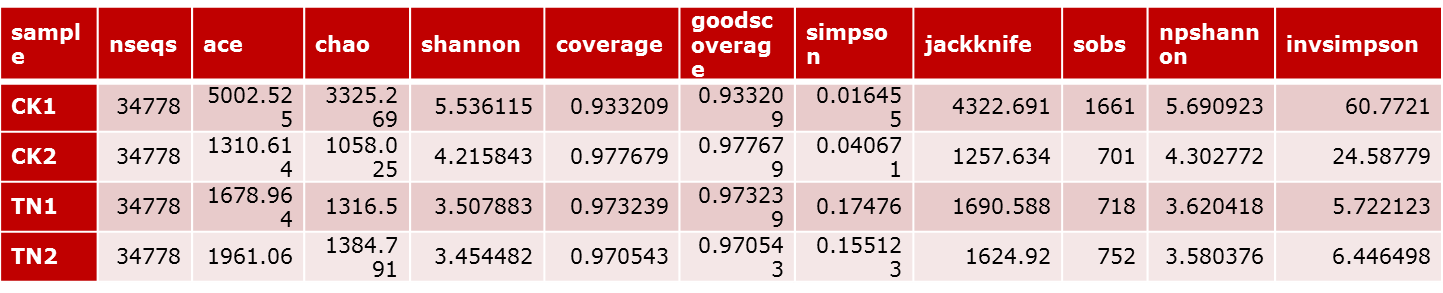
Chao1:是用chao1 算法估计群落中含OTU 数目的指数,chao1 在生态学中常用来估计物种总数,由Chao (1984) 最早提出。Chao1值越大代表物种总数越多。(菌种丰富度)
Shannon:用来估算样品中微生物的多样性指数之一。它与 Simpson 多样性指数均为常用的反映 alpha 多样性的指数。Shannon值越大,说明群落多样性越高。 (菌种多样性)
Ace:用来估计群落中含有OTU 数目的指数,由Chao 提出,是生态学中估计物种总数的常用指数之一,与Chao1 的算法不同。 (菌种丰富度)
Simpson:用来估算样品中微生物的多样性指数之一,由Edward Hugh Simpson ( 1949) 提出,在生态学中常用来定量的描述一个区域的生物多样性。Simpson 指数值越大,说明群落多样性越高。 (菌种多样性)
●多样品比较分析(Beta Diversity)
Beta Diversity是对样品间的微生物群落结构差异比较,分析方法很多。
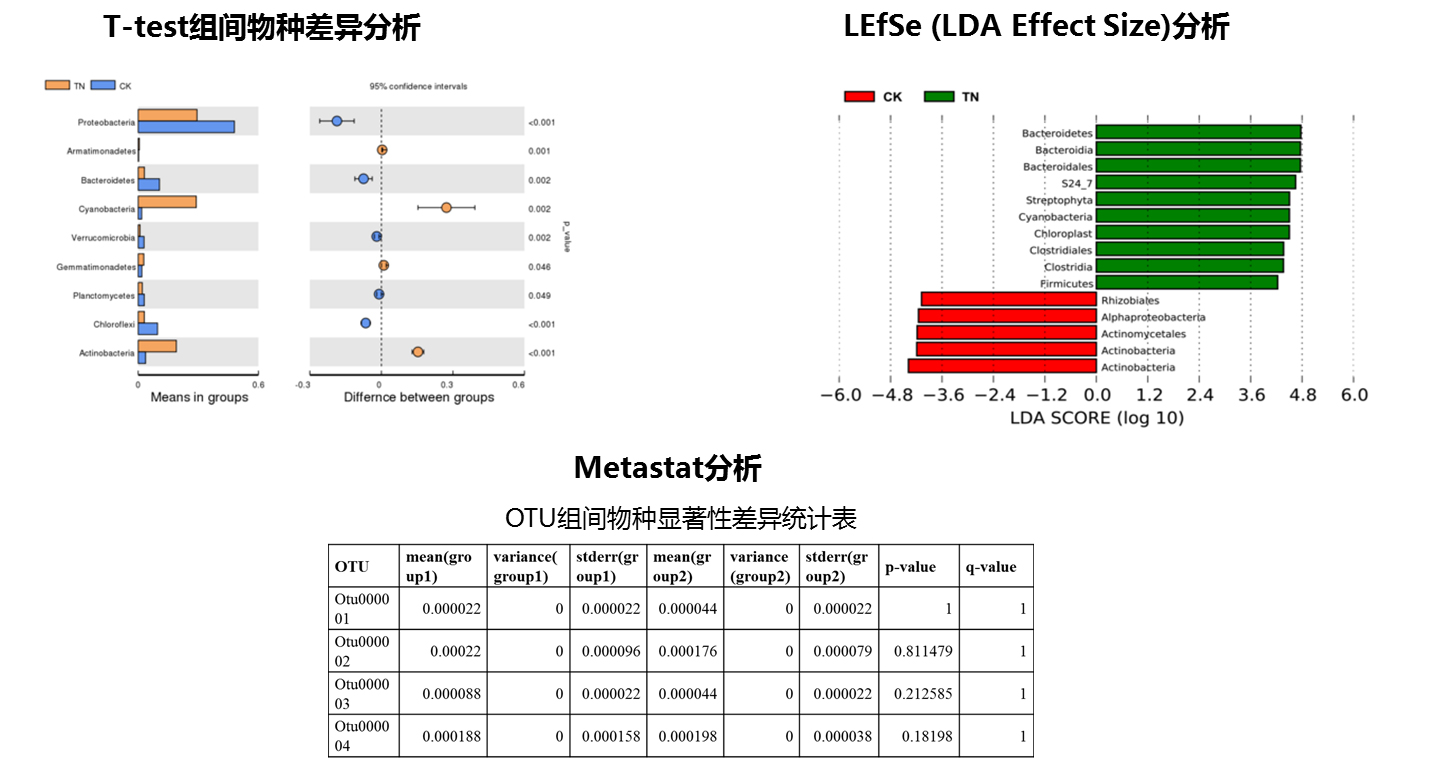
● 统计分析
针对有分组的项目,可以通过群落结构差异统计分析进行深入研究。通过统计分析,可以有针对性的找出分组间丰度变化差异显著的物种,并得到差异物种在不同分组间的富集情况,同时,可以比较组内差异和组间差异的大小,判断不同分组间的群落结构差异是否具有显著意义。
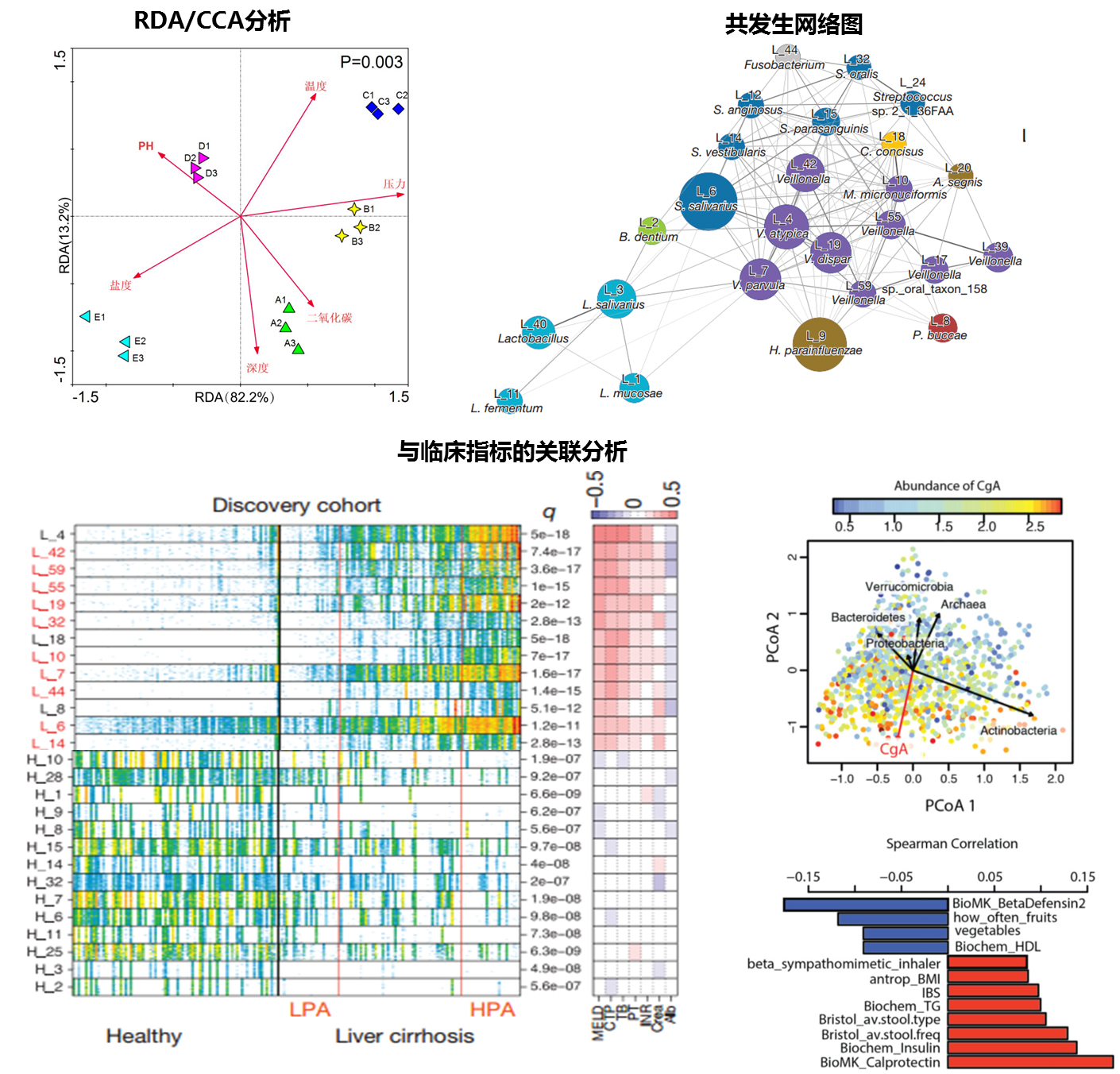
● 深入分析
中康博16S/18S/ITS测序参数

样品要求
● 提供已纯化的DNA样品(1、针对宿主的相关样品,如皮肤、口腔、呼吸道、消化道、生殖道等;2、针对环境的相关样品,如土壤、水体、空气、盐湖、沼泽等,均可进行微生物多样性检测)。
● 基因组DNA用TE溶解; 总量≥500 ng; 纯度OD260/280在1.8-2.0之间;浓度≥10ng/μL,体积≥20ul 。
● 运输要求,液氮或干冰保存寄送,北京本地客户可上门收样。
注:样本应避免各类污染和反复冻融。

